Kit FastKing RT (Cù gDNase)
Caratteristiche
■ Alta efficienza: L'Enzima FastKing RT hè mudificatu cù motivi idrofobi, cù efficienza RT più di 95%.
■ Sensibile: À menu di 1 ng mudelli ponu esse identificati cun precisione.
■ Resistenza: Capace di trascrizzione inversa di mudelli cumplessi, cù una resistenza perfetta à impurità.
■ Flessibile: A rimozione di u DNA genomicu è a trascrizione inversa sò state compie separatamente. I Primeri sò stati mischiati separatamente in un tubu, in modu flessibile per cambià altri primer.
Specificazione
Tipu: Gene mudificatu transcriptase inversa, gDNase
Procedure: In duie tappe (rimozione di DNA genomicu è RT)
Efficienza RT:> 95%
Template: 1 ng- 2 μg
Tempu di operazione: ~ 21 min
Applicazioni: U cDNA trascrittu inversu pò esse adupratu in PCR convenzionale, PCR in tempu reale, custruzzioni di libreria cDNA.
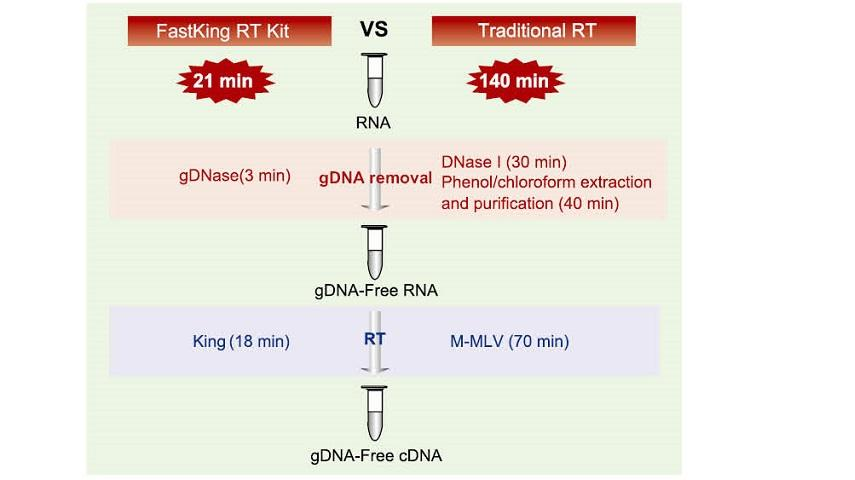
Reazione di 21 min in un tubu
Piglia solu 21 min per cumplettà a rimozione di gDNA è u prucessu di trascrizione inversa efficiente in u listessu tubu senza rimpiazzà u tubu di reazione è u prucessu di trattamentu DNase I. indipendente. Rispuntatu à u metudu tradiziunale chì richiede un'operazione in 12 passi è una reazione di 140 min, simplifica assai i passi di l'operazione è risparmia assai tempu di operazione.
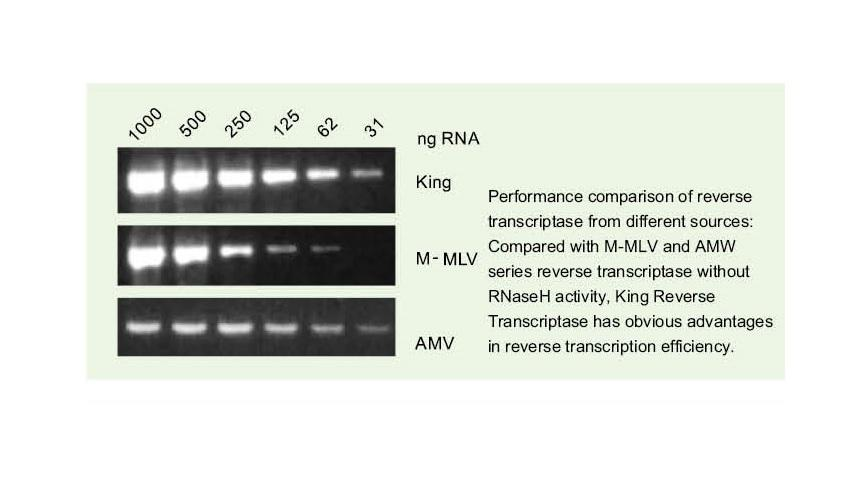
Qualità eccezziunale di King RTase
——Ultra efficienza di trascrizzione inversa
—— L'efficienza di transcrizione inversa hè più di 95%
A trascrittasi inversa generale hà una efficienza di trascrizione inversa di 40-60%, è u rendimentu di cDNA pò esse aumentatu da una quantità più alta di carica di RNA. King reverse transcriptase pò ottene una efficienza di transcrizione inversa di più di 95% per via di a so alta affinità unica per i mudelli RNA. Dunque, l'esperimenti successivi ponu esse soddisfatti senza a necessità di una grande quantità di input RNA, chì salva RNA è permette alta purezza è elevatu rendimentu di cDNA.
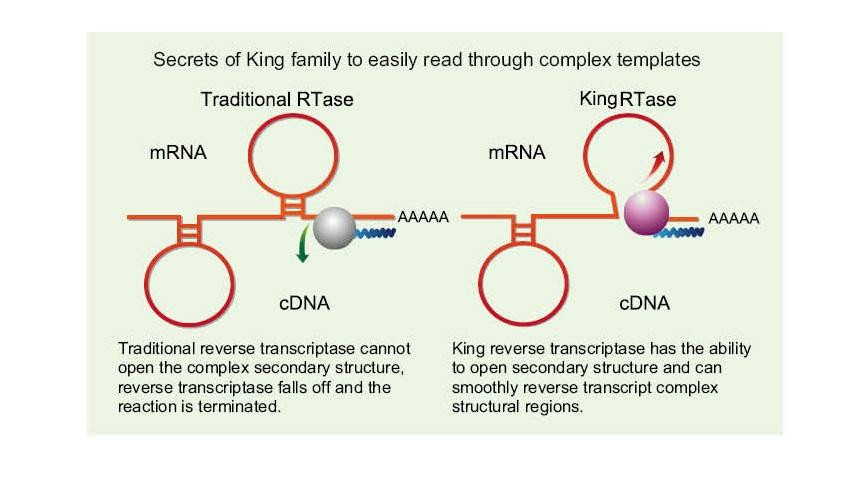
Leghje facilmente cù mudelli cumplessi
——Leghje facilmente à traversu GC altu è mudelli cumplessi
L'ARN monocatenariu hà una vasta gamma di regioni di struttura secondaria cumplessa per via di u ligame d'idrogenu trà i filamenti. A transcriptase inversa urdinaria pò purtà à a terminazione di a transcrizione inversa quandu si scontra cù una struttura secundaria cumplessa, cusì incapace di compie cù successu a sintesi di cDNA. Tuttavia, a nova generazione di King revers transcriptase hà un duminiu strutturale unicu, chì pò distrughje u ligame d'idrogenu trà i filamenti di RNA, aprendu cusì a struttura secundaria cumplessa di RNA è assicurendu a trascrizione inversa liscia.
Tutti i prudutti ponu esse persunalizati per ODM / OEM. Per i dettagli,per piacè cliccate Serviziu persunalizatu (ODM / OEM)
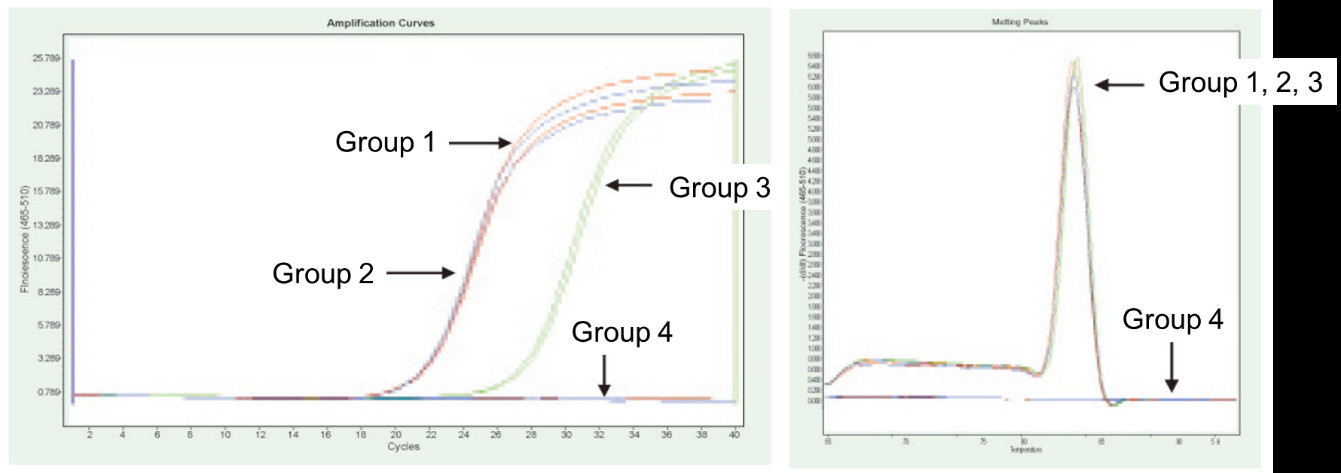 |
Gruppu 1: Trascrizione inversa senza trattamentu gDNase; Gruppu 2: Nisun trattamentu gDNase è nisuna transcrizione inversa; Gruppu 3: Trascrizione inversa dopu u trattamentu gDNase; Gruppu 4: Trattamentu gDNase senza trascrizione inversa. Metodi: Rilevazione PCR quantitativa di fluorescenza di u gene TNF-alfa (primer disegnatu nantu à l'esone cù cDNA o genomu cum'è mudellu) aduprendu 1 μg di cellula Hela RNA (cun residu di genomu) cum'è mudellu. u residu di u genomu in RNA, u gruppu 3 pò riflette accuratamente u veru livellu di spressione di TNF-alfa, u gruppu 1 hà errori in i risultati quantitativi finali per via di u residu di u genomu, è u gruppu 4 mostra chì FastKing RT Kit pò eliminà cumpletamente u DNA genomicu residuale in ARN. |
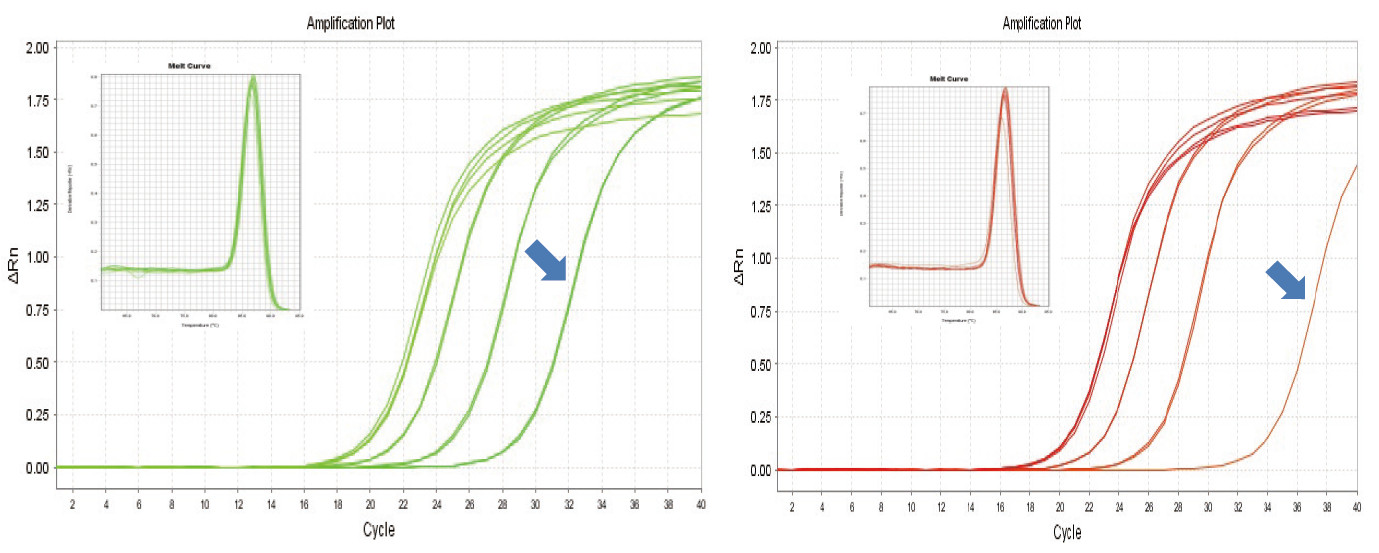 |
Figura 1. A trascrizzione inversa di u RNA di u topu hè stata fatta cù TIANGEN FastKing RT Kit (sinistra) è pruduttu pertinente di u Fornitore A (destra), allora u gene MM5 hè statu amplificatu quantitativamente aduprendu TIANGEN SuperReal PreMix Plus (SYBR Green). A curva di amplificazione è a curva di fusione sò state analizate. L'input RNA era 1000 ng, 100 ng, 10 ng è 1 ng rispettivamente. I risultati mostranu chì TIANGEN FastKing RT Kit hà un gradiente di trascrizione inversa chjaru è valore Ct bassu, è hà vantaghji evidenti per a trascrizione inversa di u mudellu di bassa abbundanza (1 ng, freccia blu). |
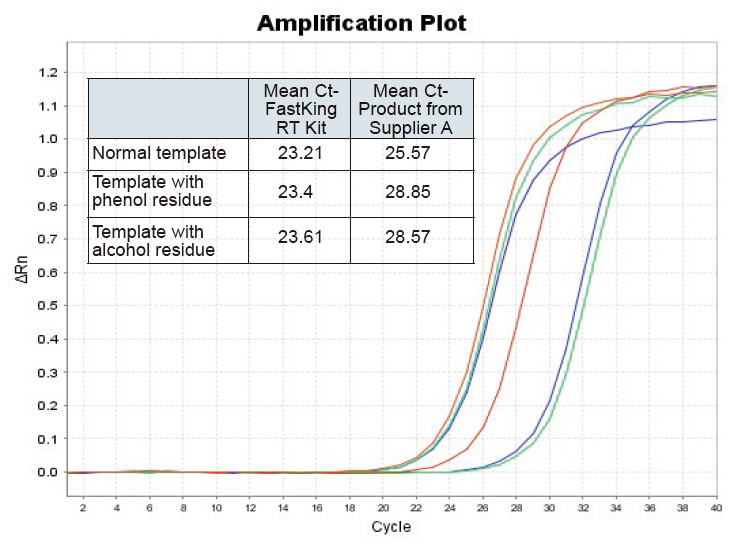 |
Figura 2. Trascrizzione inversa di mudellu RNA nurmale (rossu), mudellu cù grande residu di fenolu (verde) è mudellu cù residu di alcolu (turchinu) di topi cù TIANGEN FastKing RT Kit è produttu pertinente di u Fornitore A rispettivamente, quantificanu i geni RNC cù TIANGEN SuperReal PreMix Plus (SYBR Green), è e curve di amplificazione è i valori Ct sò stati analizzati. I risultati mostranu chì TIANGEN FastKing RT Kit hà u valore Ct quantitativu più bassu dopu a trascrizione inversa è una resistenza à u stress eccellente, è hà vantaghji evidenti per i mudelli cù residui di impurità elevati |
A-1 RNA hè degradatu
——Purificà l'ARN di alta qualità senza cuntaminazione. U materiale da u quale hè estrattu l'ARN deve esse u più frescu pussibule per prevene a degradazione di l'ARN. Analizà l'integrità di l'ARN nantu à u gel denaturatu prima di a reazione RT. Dopu l'estrazione di RNA, deve esse conservatu in 100% formamide. Se l'inibitore RNase hè adupratu, a temperatura di riscaldamentu deve esse <45 ° C, è u pH deve esse menu di 8,0, altrimenti l'inibitore libererà tuttu RNase legata. Inoltre, l'inibitore RNase deve esse aghjuntu in soluzioni chì cuntenenu ≥ 0,8 mM DTT.
A-2 RNA cuntene inibitori di reazioni di trascrizione inversa
——Inhibitori di transcrizione inversa includenu SDS, EDTA, glicerina, pirofosfatu di sodiu, spermidina, formamide, sale di guanidina, ecc. Mischjate u RNA di cuntrollu cù u campione, è paragunate u rendiment cù a reazione di RNA di cuntrollu per verificà s'ellu ci hè un inibitore. Lavate a precipitazione RNA cù 70% (v / v) etanolu per eliminà l'inibitori.
A-3 Insuffiziente annealing di primers aduprati per sintetizà u primu filu di cDNA
——Determine chì a temperatura di ricottura sia adatta per i primer aduprati in l'esperimentu. Per esameri aleatorii, si consiglia di mantene a temperatura à 25 ° C per 10 min prima di ghjunghje à a temperatura di reazione. Per primer specifici per geni (GSP), pruvate altre GSP, o cambiate à oligo (dT) o hexamer aleatoriu.
A-4 Piccola quantità di RNA iniziale
——Aumentà a quantità di RNA. Per campioni di RNA inferiori a 50 ng, da 0,1 μg a 0,5 μg di acetile BSA pò esse adupratu in a prima sintesi di cDNA
A-5 A sequenza di destinazione ùn hè micca espressa in i tessuti analizzati.
—— Pruvate altri tissuti.
A reazione A-6 PCR fiasca
——Per RT-PCR in duie tappe, u mudellu cDNA in a tappa PCR ùn pò supere 1/5 di u vulume di reazzione.
A-1 Recuit non specificu di primer è mudelli
——La fine 3'di prime ùn deve cuntene 2-3 dG o dC. Aduprate primer specifici per Gene in a prima sintesi di filamenti invece di primer aleatorii o oligo (dT). Aduprate una temperatura più alta di ricottura in i primi cicli, è dopu una temperatura più bassa di ricottura. Aduprate a polimerasa di DNA Taq hot-start per PCR per migliurà a specificità di a reazione.
A-2 Cuncepimentu scarsu di primori specifichi à u genu
—— Seguitate i listessi principii per u cuncepimentu di l'amplificazione.
A-3 RNA contaminatu da DNA genomicu
——Trattate l'ARN cù DNase di qualità PCR I. Pone in opera una reazione di cuntrollu senza trascrizione inversa per rilevà a contaminazione di l'ADN.
A-4 Formazione di primer dimer
——Design primer senza sequenze cumplementari à a fine 3 '.
A-5 Troppu altu Mg2+ cuncentrazione
——Optimize Mg2+ cuncentrazione per ogni cumbinazione di mudelli è primer
A-6 Cuntaminatu cù DNA stranieru
—— Aduprate cunsiglii resistenti à l'aerosol è enzimi UDG.
A-1 U cuntenutu di u primu pruduttu filu hè troppu altu
——Riduce a quantità di u primu pruduttu filu in u passu di reazione PCR convenzionale.
A-2 Quantità troppu alta di primer in reazione PCR
——Riduce l'input primariu.
A-3 Troppu cicli
——Optimize e cundizioni di reazione PCR è riduce u numeru di ciclu PCR.
A-4 Temperatura di ricottura troppu bassa
——Aumentà a temperatura di ricottura per prevene l'iniziazione è l'estensione non specifiche.
A-5 Amplificazione non specifica di frammenti oligonucleotidi generati da a degradazione DNase di DNA -—Estratti RNA di alta qualità per prevene a contaminazione di l'ADN.
RT-PCR hè di trascrive inversamente l'ARN in cDNA, è poi aduprà u cDNA trascrittu inversu cum'è mudellu per a reazione PCR per amplificà u frammentu target. Sceglite sia primer aleatorii, Oligo dT è primer specifici per geni secondu e cundizioni specifiche di l'esperimentu. Tutti i primer sopra ponu esse aduprati per mRNA di cellule eucariotiche corte senza struttura di forcella.
Primariu casuale: Adatta per RNA longu cù struttura di forcella, è ancu per tutti i tipi di RNA cume rRNA, mRNA, tRNA, ecc. Sò aduprati principalmente per a reazione RT-PCR di un unicu mudellu.
Oligo dT: Adatta per RNA cun coda PolyA (RNA procariota, eucariota Oligo dT rRNA è tRNA ùn anu micca coda PolyA). Perchè Oligo dT hè legatu à a coda PolyA, a qualità di i campioni di RNA hè necessaria per esse alta, è ancu una piccula quantità di degradazione riduce notevolmente a quantità di sintesi di cDNA a lunghezza intera.
Primariu specificu per u geniu: Cumplementu à a sequenza di mudelli, adattatu per situazione chì a sequenza di destinazione hè cunnisciuta.
Ci hè dui modi:
1. Metudu di riferimentu internu: In teoria, cDNA hè frammenti di DNA di lunghezze sfarente, dunque u risultatu di l'elettroforesi hè strisciu. Se l'abbundanza di RNA hè bassa, nisun produttu si presenterà in elettroforesi, ma questu ùn significa micca chì nisun pruduttu serà amplificatu da PCR. In generale, a riferenza interna pò esse aduprata per rilevà cDNA. Se a riferenza interna hà risultati, a qualità di cDNA pò esse basicamente garantita (in pochi casi, se u frammentu di u geniu target hè troppu longu, ci ponu esse eccezioni).
2. S'ellu ci hè un genu cunnisciutu amplificatu da stu mudellu, pò esse verificatu da i primori di stu genu. L'amplificazione di a riferenza interna ùn significa micca necessariamente chì ùn ci sia micca prublema cù cDNA. Perchè u riferimentu internu hà una grande abbundanza in cDNA, hè faciule da amplificà. Se cDNA hè parzialmente degradatu per vari motivi, da a prospettiva di probabilità, i risultati PCR di geni bassi di abbondanza bassa saranu assai influenzati. Mentre a riferenza interna hè sempre alta in abbundanza, l'amplificazione ùn serà probabilmente micca affettata.
Parzialmente degrade di RNA. Rileva l'integrità è purifica di RNA
U cuntenutu di RNA di e diverse spezie pò esse differente, ma in generale, l'ARN totale estrattu duveria cuntene duie bande 28S è 18S chjare in elettroforesi in gel, è a luminosità di a prima banda duveria esse duie volte più alta di quella di l'ultima. A banda 5S indica chì l'ARN hè statu degradatu, è a so luminosità hè prupurziunale à u gradu di degradazione. L'amplificazione riesciuta di a riferenza interna ùn significa micca chì ùn ci sia micca prublema cù l'ARN, perchè a riferenza interna hè in grande abbundanza, l'ARN pò esse amplificatu fintantu chì a degradazione ùn sia micca severa. U OD260/ OD280U rapportu di RNA puru misuratu da spettrofotometru deve esse trà 1,9 è 2,1. Una piccula quantità di impurità di proteine in RNA riduce u raportu. Finu chì u valore ùn hè micca troppu bassu, RT ùn serà micca affettatu. Ciò chì conta u più per RT hè l'integrità di l'ARN.
L'estensione di u genu di riferimentu internu pò indicà solu chì RT hà riesciutu, ma ùn hè micca necessariamente legatu à a qualità di u filu di cDNA. Perchè i frammenti di riferimentu internu sò generalmente chjucchi in grandezza è alti in espressione, sò più faciuli d'avè successu in trascrizione inversa. Tuttavia, a dimensione è l'espressione di u genu target varieghja da un genu à un genu. A qualità di cDNA ùn pò esse ghjudicata solu per riferenza interna in particulare per i frammenti di destinazione più lunghi di 2 kb.
Alcuni campioni anu strutture secondarie cumplesse, o anu un cuntenutu GC riccu, o sò preziosi cun poca abbundanza. In questi casi, una transcriptase inversa adatta deve esse scelta secondu a dimensione di u frammentu di destinazione è u campione. Per i mudelli RNA cun altu cuntenutu GC è struttura secundaria cumplessa, hè difficiule d'apre a struttura secundaria à bassa temperatura, o cù transcriptase inversa cumuna. Per questi mudelli, Quant Reverse Transcriptase pò esse sceltu, postu chì a so prestazione di trascrizione inversa hè ovviamente migliore di quella di a serie M-MLV reverse transcriptase, chì pò inversamente trascrivere vari mudelli di RNA in modo efficiente è trascrivere RNA in cDNA prima filu à u massimu. Quandu si usa un kit generale di trascritasi inversa, u sistema 20 μl pò trasfurmà solu inversamente 1 μg di RNA totale. Per piacè, fate attenzione à a capacità massima RT di u kit. Se u mudellu hè aghjuntu in eccessu, a trascrizzione inversa favurizarà l'ARN cù alta abbundanza. Dunque, hè megliu ùn superà a capacità massima di u sistema.
A-1 Determinate se RNA hè degradatu severamente è se RT hà successu
In generale, u mutivu di u fiascu di l'amplificazione di riferenza interna hè spessu causatu da una seria degradazione di l'ARN. Un'altra ragione pussibile hè u fallimentu di transcrizione inversa. U riferimentu internu ùn pò micca esse adupratu cum'è standard per ghjudicà a qualità di cDNA unicu filu, ma pò esse adupratu cum'è standard per ghjudicà se a trascrizzione inversa hà successu se ùn ci hè micca prublema di qualità RNA. A cosa più impurtante in u prucessu di trascrizione inversa hè di mantene una temperatura costante è un sistema di reazione costante per migliurà l'efficienza di a reazione.
A-2 Determinate se i primer per amplificà i geni di riferenza interna sò affidabili è se ci sò prublemi cù reagenti aduprati in PCR.
Per quantificazione relativa, l'ARN deve esse quantificatu prima di a trascrizione inversa, chì hè ancu necessariu in parechji kit di trascrizione inversa, per esempiu, quantificà l'ingressu RNA cum'è 1 μg. Siccomu u cDNA trascrittu inversu hè una soluzione mista, cumpresu RNA, oligo dT, enzima, dNTP, è ancu un pocu residu di DNA, a deviazione serà causata, dunque hè impussibile quantificà accuratamente u cDNA. Dunque, a quantificazione RNA hè necessaria. Cunsidirendu l'efficienza di trascrizzione inversa hè listessa trà sfarenti campioni, a quantità di cDNA ottenuta duveria esse listessa, è l'analisi quantitativa pò mustrà u paragone di i livelli di spressione di diversi geni in listessa quantità di RNA tutale. Quandu esegue una fluorescenza relativa PCR quantitativa, u cDNA quantitativu pò esse micca necessariu dopu a trascrizione inversa perchè u genu di riferimentu internu pò esse agitu cum'è riferimentu.
Hè principalmente ligatu à i geni, è a trascrizzione inversa di u frammentu longu ùn hè micca fattibile per a maiò parte di i geni. Prima, l'efficienza di a trascrizione inversa hè assai inferiore à quella di PCR. Dopu, a regione ricca in GC è a struttura secondaria di parechji geni limitanu sia a trascrizione inversa sia a PCR. Infine, a fideltà è l'efficienza di l'amplificazione di a PCR sò difficiule da garantisce in listessu tempu. In u prucessu di trascrizzione inversa, nimu ùn pò garantisce d'ottene un pezzu longu per geni bassi di copia, in particulare cù oligo dT. In quantu à 5 'UTR cù più GC, hè ancu più difficiule. Dunque, hè sempre un metudu ragiunevule per riversà a trascrizzione cù primer aleatorii, truvà i siti naturali di scissione in u frammentu di destinazione, amplificà per segmenti, è dopu eseguisce a digestione di restrizione è a ligatura. In generale, hè difficiule di amplificà direttamente frammenti più grandi di 2 kb, ma ùn hè micca sempre impussibile d'ottene: 1. Prima di tuttu, guarantisci l'integrità di RNA / mRNA, è l'estrazione TRIZOL hè preferita. U kit 2.M-MLV RT-PCR pò esse adupratu direttamente. Prolungate u tempu di ricottura è aumentate u numeru di cicculu in u prucessu di amplificazione currettamente. In alternativa, a PCR nidificata pò esse applicata, o fà una o duie reazioni prima cun denaturazione apprupriata è tempu di estensione prima di l'amplificazione PCR normale, chì pò aiutà à allargà i frammenti. Attenti à a fideltà di a polimerasa. 3. Long Taq pò esse adupratu in PCR per uttene risultati ideali. 4.Per l'applicazione di spressione di proteine, si deve applicà una polimerasi ad alta fedeltà.
Ci hè dui tipi di transcriptase inversa offerta da TIANGEN: Quant / King RTase è TIANScript M-MLV. A principal differenza trà elli hè a quantità di input di mudelli. Quant hè una trascrittasi inversa unica, chì hè differente da u M-MLV comunemente adupratu derivatu da u virus di leucemia murina Moloney. Quant hè una nova transcriptase inversa à alta efficienza ricombinante espressa da l'ingegneria Escherichia coli. Quant hè adattu per amplificà 50 ng-2 μg di RNA cù alta attività trascrizzionale inversa è altu rendimentu. Rispuntendu à MMLV ordinariu o AMV, a caratteristica più grande di Quant hè chì hà una forte affinità cù i mudelli RNA è pò riversà trascrizzione di mudelli cumplessi senza denaturazione à alta temperatura. Per mudelli cù cuntenutu GC più altu, l'efficienza inversa hè più alta. Tuttavia, sta trascrittasi inversa hà attività RNase H, chì pò influenzà a lunghezza di u pruduttu cDNA (adatta per mudelli <4,5 kb). Per a trascrizione inversa cunvinziunale, TIANScript MMLV reverse transcriptase hè cunsigliatu. Questa RTase hè un enzima mudificatu cù una attività RNase H assai debule, chì hè adatta per a sintesi longa (> 5 kb) cDNA.
A trascrizzione inversa in un passu è l'amplificazione PCR sò compie in u stessu tubu senza apre a copertura di u tubu trà sintesi di cDNA è amplificazione, chì hè utile per riduce a contaminazione. Siccomu tutti i campioni di cDNA ottenuti sò aduprati per l'amplificazione, a sensibilità hè più alta, cun un minimu di 0,01 pg di RNA totale. Per RTPCR in un passu riescitu, i primers specifici di geni sò generalmente usati per inizià a sintesi di cDNA. U metudu in duie tappe, vale à dì trascrizzione inversa è amplificazione PCR hè realizatu in duie tappe. In primu locu a trascrizione inversa hè effettuata da un mudellu RNA per ottene cDNA, è u cDNA ottenutu hè sottumessu à una o più reazioni PCR diverse. U metudu in duie tappe pò aduprà oligo (dT) o primer aleatorii per guidà a sintesi di u primu filu di cDNA, è pò inversamente trascrive tutte l'infurmazioni di mRNA da un campione specificu.
Categurie di prudutti
PERCHÉ SCEGLIÀ NOI
Dapoi a so creazione, a nostra fabbrica hà sviluppatu prudutti di prima classe mundiale cun rispettendu u principiu
di qualità prima. I nostri prudutti anu guadagnatu una reputazione eccellente in l'industria è una preziosa fiducia trà i clienti novi è vechji ..










