TIANSeq Amplificazione HiFi Mix
A valle
Amplificazione PCR di biblioteca NGS, amplificazione PCR di sequenza di 1a generazione, clonazione ad alta fedeltà, rilevazione SNP, mutazione specificu di u situ, ecc.
Caratteristiche
■ Amplificazione à alta efficienza: Assicurà u tassu di cunversione è riduce i cicli di amplificazione.
■ Preferenza Bassa: Efficienza di amplificazione equilibrata per mudelli di DNA cù cuntenuti GC% diversi.
■ Alta specificità: Cù pruprietà HotStart è forte specificità.
■ Alta fideltà: A fideltà hè 50 volte più altu ch'è Taq DNA polimerasi.
■ Alta sensibilità: L'input di u mudellu pò esse bassu à 1 pag.
Tutti i prudutti ponu esse persunalizati per ODM / OEM. Per i dettagli,per piacè cliccate Serviziu persunalizatu (ODM / OEM)
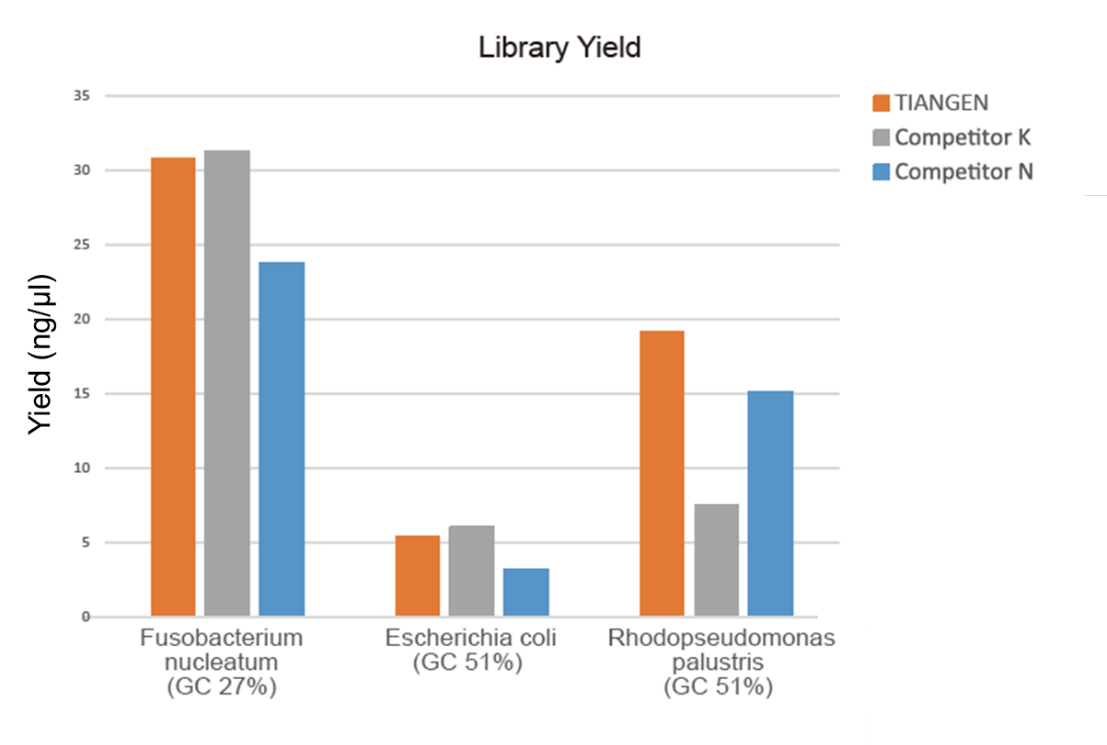 |
Figura 1. L'arricchimentu di a biblioteca di DNA genomicu cù diversi rapporti GC (ingressu genomicu 10 ng, 8 cicli di amplificazione) hè statu realizatu simultaneamente aduprendu TIANSeq HiFi Amplification Mix è HiFi enzima da u Fornitore K è N, è u rendimentu di a biblioteca hè statu rilevatu da Agilent 2100. I risultati mostranu chì TIANSeq HiFi Amplification Mix hà un elevatu rendiment di biblioteca, cù una forte universalità per diversi contenuti GC, è e prestazioni di arricchimentu di biblioteche eranu megliu cà l'altri fornitori. |
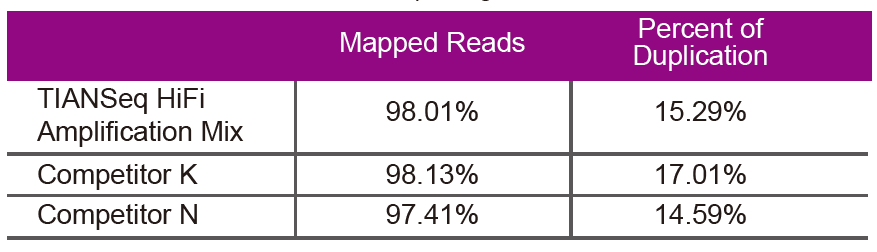 |
Dati di sequenzaAduprate TIANSeq HiFi Amplification Mix è HiFi enzyme appositamente utilizatu per l'amplificazione di libreria NGS da u Fornitore K è N per l'amplificazione di biblioteca di u stessu DNA genomicu (l'ingressu di u genomu hè 10 ng). Dopu u sequenziamentu, analizà a percentuale di copertura è di duplicazione di a biblioteca. |
Preferenza GC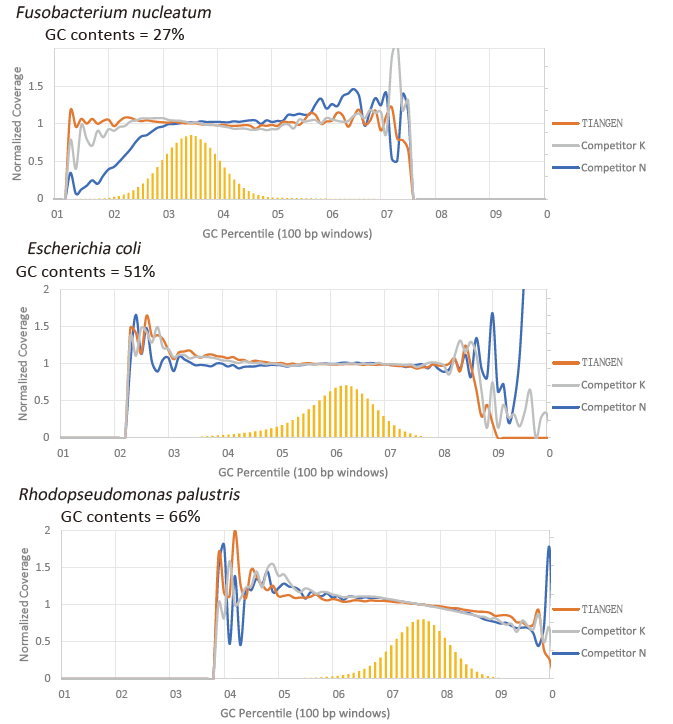 |
Figura 2. Amplificate e bibliuteche di genomu cù cuntenuti CG diversi cù TIANSeq HiFi Amplification Mix è l'HIFi da u Fornitore K è N. U risultatu mostra chì l'uniformità di a biblioteca di amplificazione TIANSeq HiFi Amplification Mix hè bona è senza preferenza GC, chì hè equivalente à i risultati di a cumpagnia K è ligeramente megliu ch'è i prudutti di a cumpagnia N. I risultati mostranu chì a cupertura di a biblioteca di amplificazione TIANSeq HiFi Amplification Mix hè alta, u tassu di duplicazione risponde à i requisiti, è e prestazioni di amplificazione di a biblioteca sò equivalenti à quella di i cuncurrenti. |
Oghje, a tecnulugia di sequenziazione à elevata capacità hè basata principalmente nantu à a tecnulugia di sequenziazione di prossima generazione. Cum'è a lunghezza di lettura di a tecnulugia di sequenziazione di a prossima generazione hè limitata, duvemu rompe a sequenza di lunghezza intera in piccule librerie di frammenti per sequenziallu. Sicondu i bisogni di e diverse sperienze di sequenziazione, di solitu sceglimu una sequenziazione unifundita o una sequenziazione doppia. Attualmente i frammenti di DNA di a biblioteca di sequenziazione di a prossima generazione sò generalmente distribuiti in a gamma di 200-800 bp.

a) L'ADN hè di qualità scarsa è cuntene inibitori. Aduprate campioni di DNA di alta qualità per evità l'inibizione di l'attività enzimatica.
b) A quantità di campione di DNA hè insufficiente quandu si usa un metudu senza PCR per custruisce una biblioteca di DNA. Quandu l'entrata di u DNA frammentatu supera 50 ng, u flussu di travagliu senza PCR pò esse effettuatu selettivamente durante u prucessu di custruzzioni di a biblioteca. Se u numeru di copia di a biblioteca hè troppu bassu per esse sequenziatu direttamente, a biblioteca di DNA pò esse amplificata da PCR dopu a ligatura di l'adattatore.
c) A contaminazione di l'ARN porta à una quantificazione iniziale di DNA imprecisa A contaminazione di l'ARN pò esiste in u prucessu di purificazione di DNA genomicu, chì pò purtà à una quantificazione di DNA imprecisa è à carica insufficiente di DNA durante a custruzzione di a biblioteca. RNA pò esse eliminatu trattendu cù RNase.
A-1
a) I picculi frammenti (60 bp-120 bp) apparsu I picculi frammenti sò generalmente frammenti di adattatori o dimeri furmati da adattatori. A purificazione cù perline magnetiche Agencourt AMPure XP pò rimuovere efficacemente questi frammenti di adattatori è assicurà una sequenza di qualità.
b) Grandi frammenti cumpariscenu in a biblioteca dopu l'amplificazione PCR A dimensione di u fragmentu di DNA di biblioteca aumenterà di 120 bp dopu chì l'adattatore sia ligatu. Se u frammentu di DNA aumenta di più di 120 bp dopu a ligatura di l'adattatore, puderia esse causatu da amplificazione di frammenti anormali di amplificazione PCR eccessiva. Riduce u numeru di cicli di PCR pò prevene a situazione.
c) Dimensione anormale di frammenti di DNA di biblioteca dopu a ligatura di l'adattatore A lunghezza di l'adattatore in questu kit hè 60 bp. Quandu e duie estremità di u frammentu sò ligate à l'adattatori, a lunghezza aumenterà solu di 120 bp. Quandu aduprate un adattatore diversu da quellu furnitu da stu kit, cuntattate u fornitore per furnisce informazioni pertinenti cume a lunghezza di l'adattatore. Assicuratevi chì u flussu di travagliu è l'operazione di l'esperimentu seguenu i passi descritti in u manuale.
d) Dimensione anormale di u fragmentu di DNA prima di a ligatura di l'adattatore A ragione di stu prublema pò esse causata da cundizioni di reazione sbagliate durante a frammentazione di DNA. Diversi tempi di reazione devenu esse aduprati per input di DNA diversi. Se l'entrata di DNA hè più di 10 ng, vi raccomandemu di sceglie u tempu di reazione di 12 min cum'è u tempu di partenza per l'ottimisazione, è a dimensione di u frammentu pruduttu in questu mumentu hè principalmente in u range di 300-500 bp. L'utilizatori ponu aumentà o diminuisce a lunghezza di frammenti di DNA per 2-4 min secondu e so esigenze per ottimizà i frammenti di DNA cù a dimensione necessaria.
A-2
a) U tempu di frammentazione ùn hè micca ottimizatu Se u DNA frammentatu hè troppu chjucu o troppu grande, per piacè riferitevi à e Linee Guida per a Selezzione di u Tempu di Frammentazione furnite in l'istruzzioni per determinà u tempu di reazione, è utilizate stu puntu di tempu cum'è un cuntrollu, aghjustate ancu un sistema di reazzione per allungà o accurtà 3 min per fà un aghjustamentu più precisu nantu à u tempu di frammentazione.
A-3
Distribuzione anormale di dimensioni di DNA dopu trattamentu di frammentazione
a) Metudu di scongelamentu scorrettu di reagente di frammentazione, o u reagente ùn hè micca cumpletamente mischiatu dopu à u scongelamentu. Scongelate u reattivu 5 × Frammentazione Enzyme Mix nantu à u ghjacciu. Una volta scongelatu, mischjà u reagente uniformemente sfugliendu delicatamente u fondu di u tubu. Ùn vorticiate micca u reagente!
b) U campionu d'entrata di DNA cuntene EDTA o altri inquinanti L'esaurimentu di ioni di sale è agenti chelanti in u passu di purificazione di l'ADN hè particularmente impurtante per u successu di l'esperimentu. Se l'ADN hè scioltu in 1 × TE, aduprate u metudu furnitu in l'istruzzioni per fà a frammentazione. Se a concentrazione EDTA in a soluzione hè incerta, si raccomanda di purificare l'ADN e dissolverlo in acqua deionizzata per una reazione successiva.
c) Quantificazione di DNA iniziale imprecisa A dimensione di u DNA frammentatu hè strettamente ligata à a quantità di input di DNA. Prima di trattamentu di frammentazione, una quantificazione precisa di DNA cù Qubit, Picogreen è altri metudi hè essenziale per determinà a quantità esatta di DNA in u sistema di reazione.
d) A preparazione di u sistema di reazione ùn seguita micca l'istruzzioni A preparazione di u sistema di reazione frammentatu deve esse effettuata nantu à u ghiacciu strettamente secondu e istruzioni. Per assicurà u megliu effettu, tutti i cumpunenti di a reazione devenu esse posti nantu à u ghjacciu è a preparazione di u sistema di reazione deve esse effettuata dopu un raffreddamentu cumpletu. Dopu chì a preparazione hè finita, per piacè lampate o pipette per mischjà accuratamente. Ùn vortici!
1. Un metudu di mischju impropriu (vortici, oscillazione viulente, ecc.) Causerà una distribuzione anormale di i frammenti di biblioteca (cum'è mostratu in a figura seguente), affettendu cusì a qualità di a biblioteca. Dunque, quandu preparate a soluzione di reazione di Frammentazione Mix, per piacè pipettate delicatamente su e giù per mischjà, o aduprate a punta di u dittu per lampà è mischjà uniformemente. Attenti à ùn mischjassi cun vortici.

2. Un DNA di alta purezza deve esse adupratu per a custruzzione di bibliuteche
■ Bona integrità di l'ADN: a banda di l'elettroforesi hè più di 30 kb, senza coda
■ OD260 / 230:> 1.5
■ OD260 / 280: 1.7-1.9
3. A quantità di input di DNA deve esse accurata Si suggerisce di aduprà metudi Qubit è PicoGreen per quantificà u DNA, piuttostu chè Nanodrop.
4. U cuntenutu di EDTA in soluzione di DNA deve esse determinatu EDTA hà una grande influenza nantu à a reazione di frammentazione. Se u cuntenutu di EDTA hè altu, a purificazione di l'ADN deve esse effettuata prima di u test successivo.
5. A suluzione di reazzione di frammentazione deve esse preparata nantu à u ghjacciu U prucessu di frammentazione hè sensibile à a temperatura è à u tempu di a reazione (in particulare dopu avè aghjuntu un amplificatore). Per assicurà l'accuratezza di u tempu di reazione, per piacè preparate u sistema di reazione nantu à u ghjacciu.
6. U tempu di reazione di frammentazione deve esse precisu U tempu di reazione di u passu di frammentazione hà da influenzà direttamente a dimensione di i prudutti di frammentu, affettendu cusì a distribuzione di dimensioni di i frammenti di DNA in a biblioteca.
1. Chì tippu di campione hè applicabile à stu kit?
U tippu di campione applicabile di questu kit pò esse RNA totale o mRNA purificatu cun bona integrità RNA. Se RNA totale hè adupratu per custruisce a libreria, hè raccomandatu di aduprà u kit di deprezzamentu di rRNA (Cat # 4992363/4992364/4992391) per rimuovere prima l'RRNA.
2. Si ponu aduprà campioni FFPE per custruisce una biblioteca cù stu kit?
U mRNA in i campioni FFPE serà degradatu in una certa misura, cù una relativa integrità scarsa. Quandu si usa stu kit per a custruzzione di a biblioteca, si consiglia di ottimizà u tempu di frammentazione (accurtà u tempu di frammentazione o ùn eseguisce micca a frammentazione).
3. Aduprendu u passu di selezzione di dimensione furnitu in u manuale di u pruduttu, chì puderebbe fà chì u segmentu inseritu apparisce una leggera deviazione?
A selezzione di taglia serà effettuata in stretta cunfurmazione cù u passu di selezzione di taglia in stu manuale di pruduttu. Se ci hè una deviazione, a ragione pò esse chì e perle magnetiche ùn sò micca equilibrate à a temperatura di l'ambiente o ùn sò micca cumpletamente mescolate, a pipetta ùn hè micca precisa o u liquidu hè restatu in punta. Hè raccomandatu di aduprà i cunsiglii cù bassa adsorbimentu per u sperimentu.
4. Selezzione di adattatori in a custruzzione di bibliuteche
U kit di custruzzione di a biblioteca ùn cuntene micca reagent adattatore, è si consiglia di aduprà stu kit cun TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378).
5. QC di a biblioteca
Rilevazione quantitativa di biblioteca: Qubit è qPCR sò aduprati per determinà a concentrazione di massa è a concentrazione molare di a biblioteca rispettivamente. L'operazione hè strettamente conforme à u manuale di u pruduttu. A concentrazione di a biblioteca risponde generalmente à i requisiti di sequenza NGS. Rilevazione di a gamma di distribuzione di biblioteca: Utilizà Agilent 2100 Bioanalyzer per rilevà a gamma di distribuzione di biblioteca.
6. Scelta di u numeru di ciclu di amplificazione
Sicondu l'istruzzioni, u numeru di cicli di PCR hè 6-12, è u numeru di cicli di PCR necessarii deve esse sceltu secondu l'input campione. In biblioteche ad alto rendimento, l'amplificazione eccessiva accade di solitu in gradi variabili, chì si manifesta da un piccu leggermente più grande dopu u piccu di a gamma di destinazione in a rilevazione di Agilent 2100 Bioanalyzer, o a concentrazione rilevata di Qubit è inferiore a quella di qPCR. Mild over amplification hè un fenomenu nurmale, chì ùn affetta micca a sequenza di biblioteche è l'analisi di dati successivi.
7. Spikes apparisce in u prufilu di rilevazione di Agilent 2100 Bioanalyzer
L'apparizione di punte in a rilevazione Agilent 2100 Bioanalyzer hè per via di a frammentazione irregulare di campioni, induve ci saranu più frammenti in certa dimensione, è questu diventerà più evidente dopu l'arricchimentu PCR. In questu casu, hè suggeritu di ùn fà micca a selezzione di dimensione, vale à dì mette a cundizione di frammentazione à 94 ° C per 15 min incubati, induve a distribuzione di i frammenti hè chjuca è cuncintrata, è l'umogeneità pò esse migliurata.
Categurie di prudutti
PERCHÉ SCEGLIÀ NOI
Dapoi a so creazione, a nostra fabbrica hà sviluppatu prudutti di prima classe mundiale cun rispettendu u principiu
di qualità prima. I nostri prudutti anu guadagnatu una reputazione eccellente in l'industria è una preziosa fiducia trà i clienti novi è vechji ..








